[THESE] Conception conjointe de masques de phase et d’algorithmes pour la microscopie de localisation super-résolue (PALM) dans les milieux biologiques
- Recherche
- Thèse et post-doc
- Laboratoire Charles Fabry (Palaiseau)
- Laboratoire Charles Fabry, Imagerie et Information
Contexte
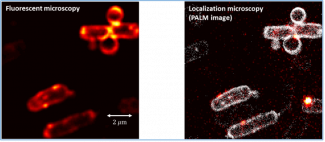
La microscopie de localisation photo-activée (PALM) permet, grâce à des microscopes performants et des techniques de traitement d'image avancées, d'estimer la position d'une particule fluorescente avec une précision bien supérieure à la limite de diffraction et ainsi de reconstruire des images "super-résolues" ayant une résolution nanométrique [1,2] (Fig. 1). Les biologistes souhaitent aujourd'hui imager des milieux biologiques épais, et la profondeur de champ (PdC) naturelle des microscopes PALM standards n'est pas suffisante. Il est donc nécessaire d'augmenter cette profondeur de champ [3,4] et, dans certaines applications, d'estimer la position tridimensionnelle de la particule dans le volume de l'échantillon [5-7].
Figure 1: (gauche) Image de bactérie par microscopie de fluorescence. Résolution ~200 nm (limite de diffraction). (droite) Image du même échantillon avec un microscope PALM. Résolution: 10 nm. Crédit: Antony Lee, Univ. of California, Berkeley.
La PdC peut être augmentée en plaçant des masques de phase optimisés dans le diaphragme d'ouverture du microscope afin de contrôler la variation de la fonction d'étalement du point (FEP, ou PSF en anglais pour Point Spread Function) avec la profondeur, et en appliquant des algorithmes de traitement adaptés [3,4]. On voit sur la figure 2 qu'avec un tel masque, la PSF du microscope reste contrastée et la précision de localisation varie très peu avec la défocalisation. Cette nouvelle technique augmente considérablement la profondeur d'exploration des méthodes d'imagerie de localisation de molécules uniques, qui est une problématique fondamentale dans l'étude des milieux biologiques.
Figure 2: (gauche) Microscope avec masque de phase. (milieu) PSF à différentes profondeurs, sans masque et avec masque optimal. (droite) Précision de localisation sans masque (bleu) et avec masque de phase optimal (rouge) en fonction de la défocalisation.
Le projet
Il est clair que les masques optimaux dépendent des exigences spécifiques de chaque application. En effet, leur optimisation est un compromis entre la précision de localisation (en 2D ou 3D), la profondeur d'exploration et le bilan photonique (qui est lié à la fréquence d'imagerie et aux problèmes de phototoxicité). Aujourd’hui, les biologistes doivent être capables de concevoir le bon masque pour chaque application.
L'objectif de ce projet de thèse est de répondre à cette problématique en développant un cadre complet d'optimisation des masques de phase qui prenne pleinement en compte les paramètres et contraintes liés à chaque application d'imagerie PALM. Cela conduira à revisiter les critères d'optimisation des masques d'amélioration de la PdC et d'estimation 3D, et ainsi à concevoir de nouveaux types de masques plus adaptés aux conditions réelles d'imagerie.
Au cours de ce projet, les masques seront optimisés numériquement et validés par des mesures réelles effectuées en collaboration avec l’équipe du laboratoire LP2N en Optique et Biologie, qui met en œuvre des microscopes PALM et est reconnue mondialement pour son expertise dans le développement de la microscopie à très haute résolution pour l’imagerie biologique. Nous avons récemment mis en œuvre sur ces microscopes des masques optimaux de type « binaire annulaire » et avons la possibilité d’acquérir des images à profondeur de champ augmentée pour différentes applications. Grâce à cette approche interdisciplinaire, des améliorations décisives de l'imagerie PALM appliquée aux sciences de la vie et à la biologie moléculaire sont attendues.
Le plan de travail
Ce projet de doctorat est basé sur deux blocs étroitement interconnectés.
1. Développement et validation d'une méthodologie d'optimisation des masques de phase adaptée aux besoins applicatifs, tels que :
- Compromis entre la précision 2D et la profondeur de champ ou la précision 3D.
- Budget photonique : temps d'acquisition disponible, performances du fluorophore, résistance de l'échantillon à la phototoxicité.
- Compromis entre les performances de détection des pics et la précision de la localisation sub-pixel.
L'optimisation du masque se fera en maximisant le taux de détection des pics et la borne de Cramer Rao sur la précision de la localisation [3,6,7]. Un paramètre important à étudier sera la paramétrisation appropriée de la fonction de phase des masques (phase binaire, décomposition sur des bases polynomiales telles que Zernike, Laguerre-Gauss, ...).
2. Conception d'une chaîne de traitement complète pour les images PALM réelles dans un contexte d'imagerie à PdC étendue et en 3D. En particulier :
- Conception de nouvelles méthodes pour la pré-détection des pics en présence d'aberrations et de fonds non uniformes. Les concepts et méthodes de la théorie de la détection et machine learning seront utilisés pour traiter les arrière-plans complexes.
- Concevoir des algorithmes pour la localisation précise de particules en 2D lorsque la PSF du microscope n'est que partiellement connue et varie avec la profondeur.
- Concevoir des algorithmes pour l'estimation de la position 3D des particules avec des masques de phase optimisés. Les algorithmes appropriés dépendront de la façon dont le PSF varie avec la défocalisation, et donc du type de masque.
Les travaux sur ces deux blocs seront menés en parallèle, en augmentant progressivement la complexité et le réalisme des milieux simulés, des masques optimaux et des algorithmes de traitement. Les résultats de ces études conduiront à revisiter les critères d'optimisation des masques d'amélioration de la PdC et d'estimation 3D, et ainsi à concevoir de nouveaux types de masques plus adaptés aux conditions réelles d'imagerie. Les résultats seront validés expérimentalement par des mesures réelles effectuées sur des microscopes PALM, et les retours de ces expériences permettront d’améliorer les modèles utilisés pour l’optimisation des masques.
Profil recherché
Ce projet fait appel à des compétences interdisciplinaires et requiert une motivation pour la modélisation optique et le traitement des images. Le ou la candidat·e retenu·e possèdera idéalement des compétences dans ces deux domaines. Cependant, ce projet convient aussi parfaitement à une personne compétente en physique et désireuse de développer ses connaissances en traitement d'images, ou bien compétente en traitement d’image et souhaitant améliorer ses connaissances en modélisation optique.
Références
[1] E. Betzig et al. “Imaging intracellular fluorescent proteins at nanometer resolution” Science 313, 1642–1645 (2006).
[2] F. Diaz et al., “Real-time increase in depth of field of an uncooled thermal camera using several phase-mask technologies”, Opt. Lett. 36 (3), 418-420 (2011).
[3] R. Falcón, F. Goudail, C. Kulcsár, H. Sauer, “Performance limits of binary annular phase masks codesigned for depth-of-field extension,” Opt. Eng. 56(6), 065104 (2017).
[4] O. Lévêque et al, “Co-designed annular binary phase masks for depth-of-field extension in single-molecule localization microscopy”, Opt. Express 28, 32426-32446 (2020).
[5] S. R. Pavani et al. ”Three-dimensional, single-molecule fluorescence imaging beyond the diffraction limit by using a double-helix point spread function”, PNAS, 106, 2995--2999, (2009).
[6] Y. Shechtman, S.J. Sahl, A.S.Backer and W.E. Moerner, “Optimal point spread function design for 3D imaging“, Physical Review Letters 113, 133902 (2014).
[7] P. Bon, J. Linarès-Loyez, M. Feyeux, K. Alessandri, B. Lounis, P.Nassoy, L. Cognet, “Self-interference 3D super-resolution microscopy for deep tissue investigations”, Nature Methods 15, 449–454 (2018)
Contacts
François GOUDAIL - francois.goudail@institutoptique.fr
Caroline KULCSÁR - caroline.kulcsar@institutoptique.fr